华南师范大学环境学院应光国教授团队耐药小组高方舟、何良英、白红等有关养殖环境人体肠道抗性组的研究成果发表在环境领域期刊Journal of Hazardous Materials(DOI:10.1016/j.jhazmat.2023.133082)。

图文摘要
成果简介
抗生素耐药性基因(ARGs)在牲畜养殖环境中普遍存在,但其对当地居民肠道微生物和抗性组的影响知之甚少。本研究基于宏基因组测序技术调查了养猪村居民的肠道微生物组和抗性组,及其环境关联,并与非养殖村居民进行比较研究。结果显示:相比非养殖村的居民,养殖村居民的肠道微生物组更不健康,携带多样的病原菌和ARGs。人肠道中的抗性组组成与猪粪便和空气中的组成更相似。可移动基因元件与肠道抗性组关联紧密。一些质粒携带的ARGs在肠道和环境样本中具有相似的基因环境。人肠道中组装获得了47个携带ARGs的细菌科,其中肠杆菌科细菌具有最高耐药和致病威胁。肠道和环境样本(主要在猪粪便和空气中)中检出的同种ARGs在同一个细菌属中序列保守。
环境意义
环境抗微生物耐药性(AMR)可通过多种暴露途径传播进入人体。畜禽养殖场是AMR发展、传播和流行的热区,可导致ARGs转移到当地居民,并塑造肠道抗性组,其引起的细菌耐药问题可能影响当地居民的身体健康。
全文速览
引言
抗生素耐药是全球性的公共健康问题。联合国粮农组织、环境署、世界卫生组织、世界动物卫生组织共同提出,须基于“One-Health”理念制定遏制微生物耐药行动计划。我国是养殖大国,养殖过程使用的抗生素占全国总使用量的一半以上,导致养殖环境抗生素耐药问题严重。《遏制微生物耐药国家行动计划(2022-2025年)》强调,要深入开展养殖环境中微生物耐药机制和传播机制研究。我国养殖从业人员总数超1000万,其中散户约占60%。相比于规模化养殖,散户养殖一般密集分布在农村地区,缺少先进的养殖技术和管理理念,处理养殖粪污的措施较为原始,卫生条件较差,环境中的耐药细菌和抗性基因可能更易定殖在这些养殖人群。然而,目前仍不清楚养殖对周边居民肠道微生物组和抗性组的影响,哪些养殖源抗性基因会可能传播到人体,以及它们的水平转移潜力如何。
研究方法
本研究基于宏基因组测序研究我国广西某养猪村居民的肠道微生物组和抗性组,并于临近非养殖村居民进行比较;通过分析可移动遗传元件与抗性组的关系,分析肠道抗性组的转移潜力;通过微生物溯源和序列比对的方法研究肠道ARGs的潜在环境来源(包括猪粪便、土壤、地下水、地表水和空气);基于宏基因组组装的方法研究环境和肠道样品中ARGs的宿主细菌和序列保守型。
主要结果
研究发现,养殖村和非养殖村居民肠道的微生物组成和功能组成不存在显著差异,但养殖村居民肠道的抗生素耐药风险更高、肠道微生物组健康指数更低、致病菌的丰度与多样性更高。
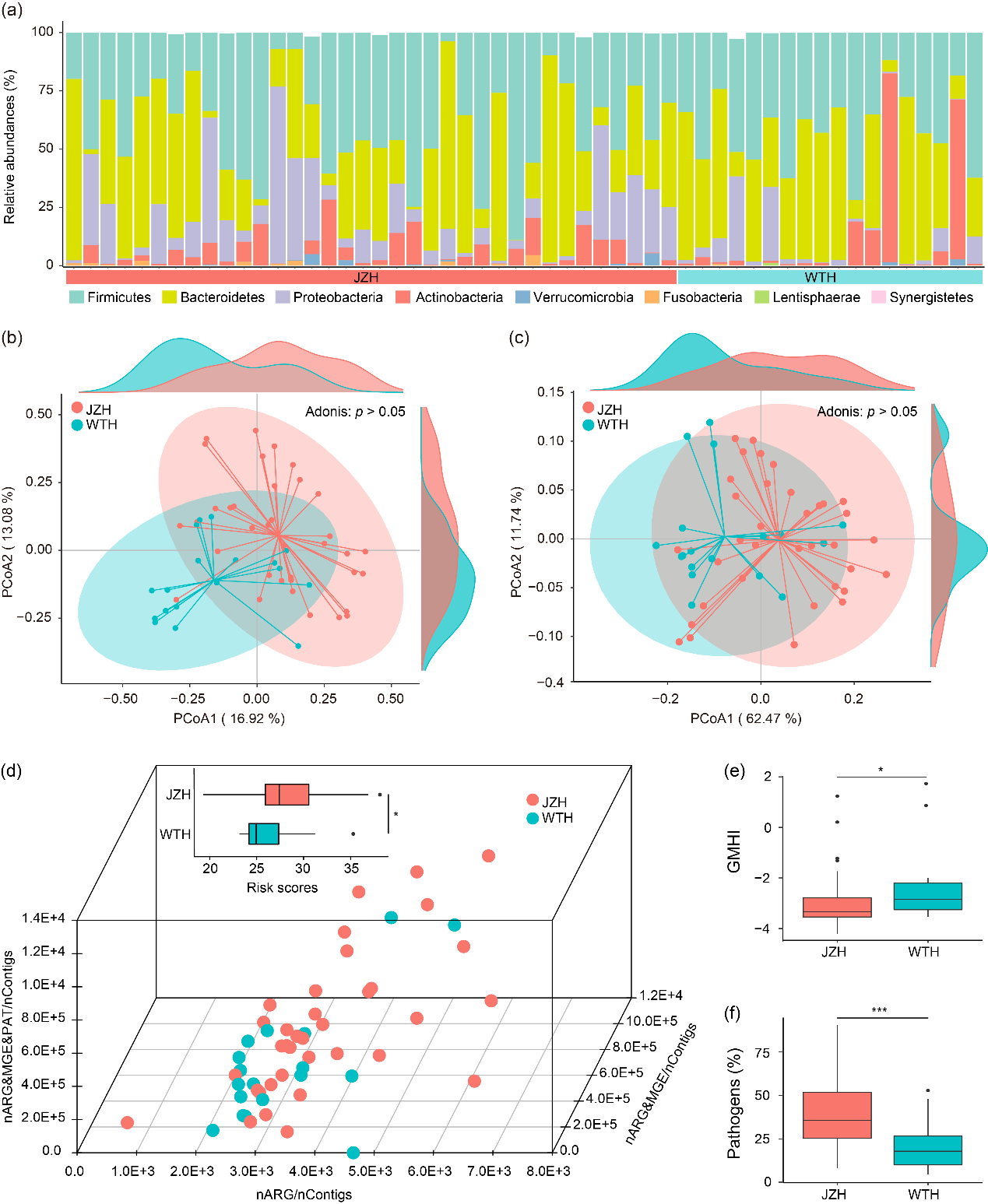
Fig. 1. Overview of gut microbiome in the human gut. (a) Relative abundance of each bacterial phylum; (b) Variations of bacterial composition in human gut samples; (c) Variations of microbial function in human gut samples; (d) Antibiotic resistance risks; (e) The Gut Microbiome Health Index; and (f) Relative abundance of total pathogens.
养殖村居民肠道携带更多的ARGs种类,其中144种未在非养殖村居民中检出。相比而言,非养殖村居民特有的ARGs只有24种。此外,在所有人体样品中共发现22种Risk rank I的ARGs,其中7种只在养殖村居民中检出。两类人群共有的ARGs中,23种在养殖村居民中的丰度显著高于非养殖村居民。养殖村居民肠道与空气(171种)、猪粪(85种)共有的ARGs种类最多,而与水土介质共有的种类较少。
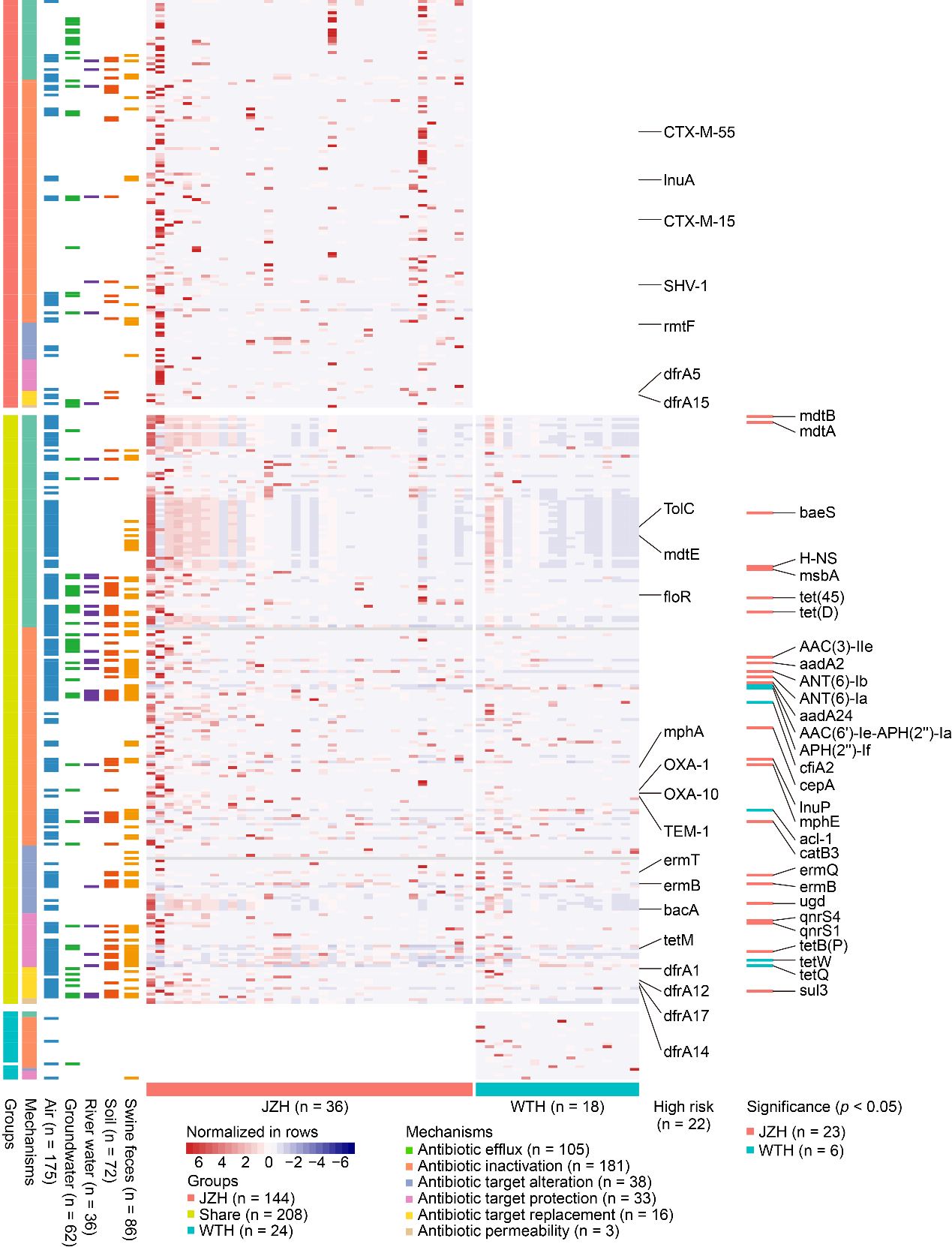
Fig. 2. Antibiotic resistome in the human gut. Colored stripes in left seven columns represent presence in groups. The ARGs colored in grey in the heatmap are detected but not quantified in human gut samples.
养殖村居民肠道中检出大量的可移动遗传元件,其种类和丰度显著高于非养殖村居民,插入序列、整合子和质粒的总丰度与肠道抗性组呈线性相关。
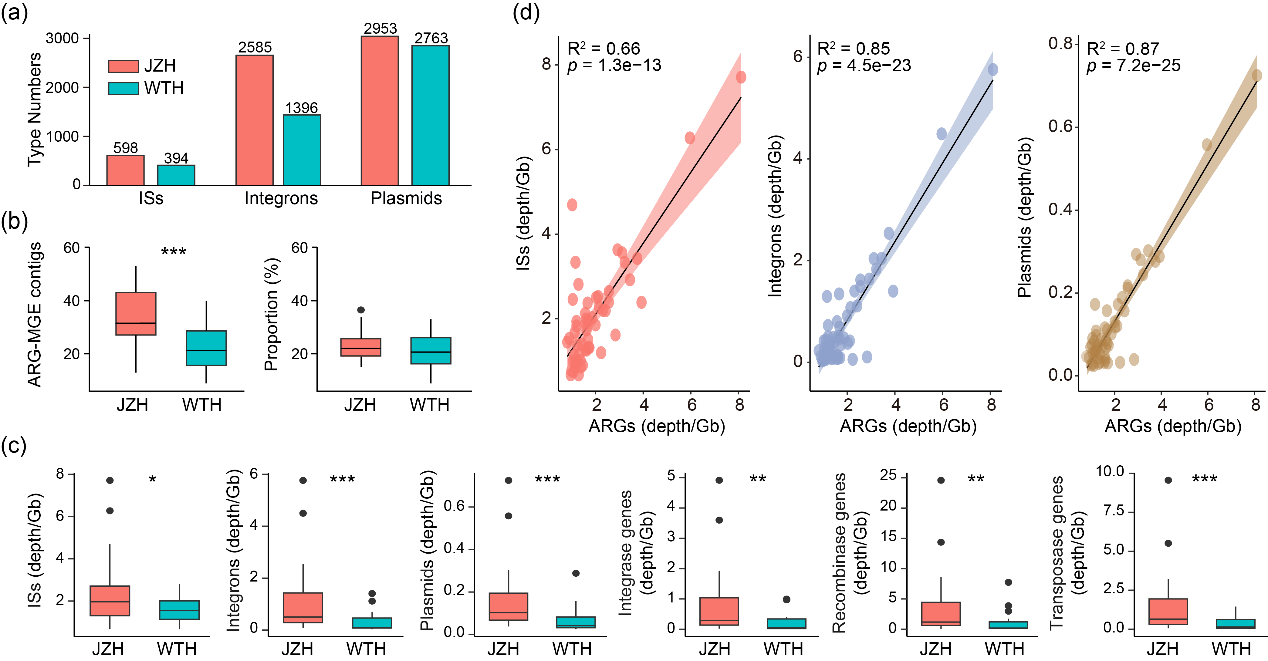
Fig. 3. Profiles of MGEs in the human gut. (a) Detected numbers of insertion sequence, integron and plasmid types; (b) Numbers of ARG-MGE contigs and their proportions in total ARGs-carrying contigs; (c) Relative abundance of each MGE class; and (d) linear regression of each MGE class and antibiotic resistome in the human gut.
养殖村居民肠道平均检出7.6条质粒序列,是非养殖村居民的1.5倍,一些序列携带多种ARGs。同源性分析发现,养殖村居民肠道中的质粒序列与环境中的序列相似性较高,可能来源于环境。然而,所有环境样品中均为发现与非养殖村居民肠道质粒序列同源性较高的序列,这更加说明养殖村居民的质粒序列来源于养殖环境。
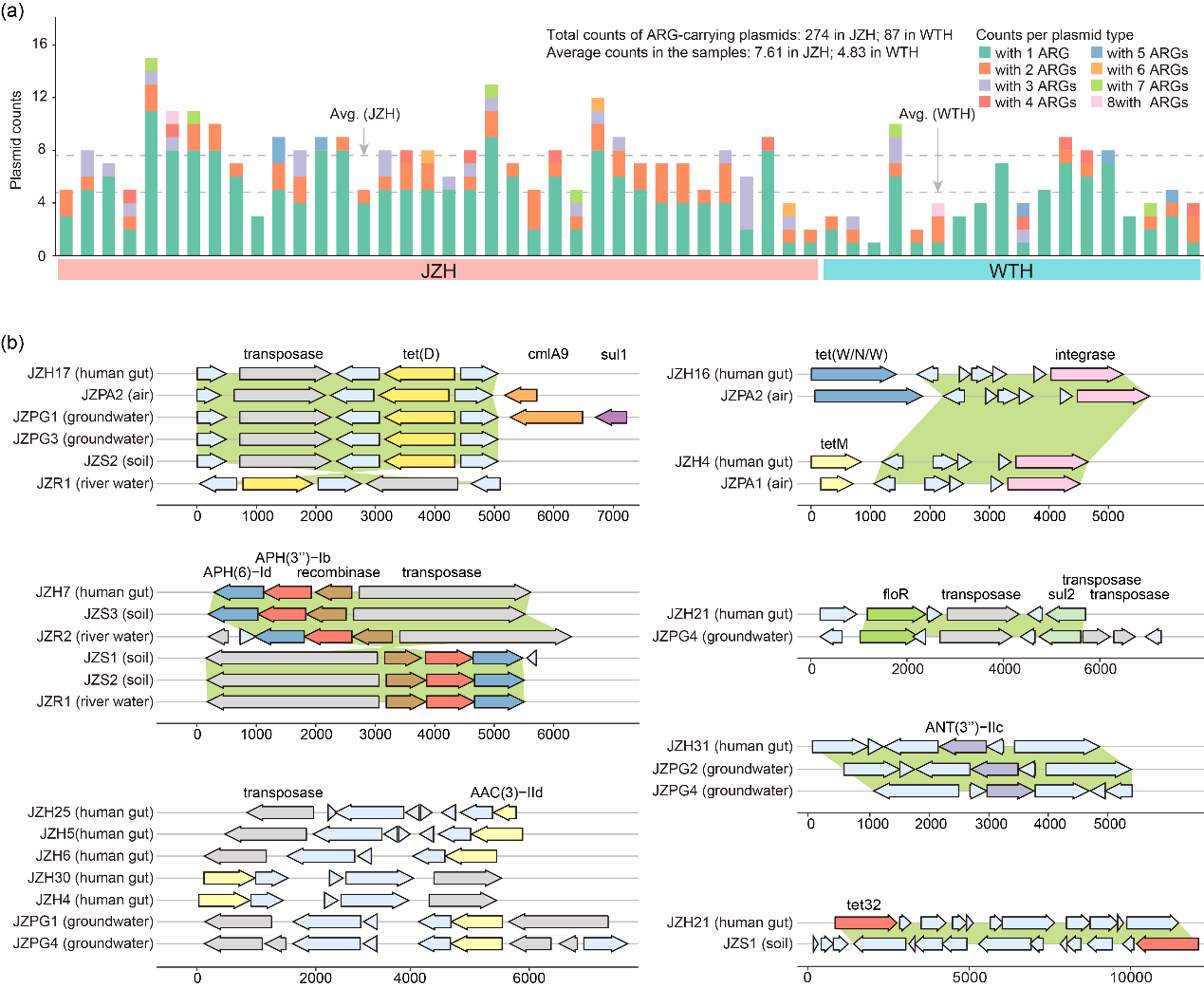
Fig. 4. Plasmid-borne ARGs in the human gut with homologies in environments. (a) Numbers of ARGs-carrying plasmids (fragments); and (b) Genetic contexts of the plasmid-borne ARGs in guts and environments.
所有样品中共获得3490个基因组,其中56%来自养殖村人肠道、26%来自非养殖村人肠道、8%来自猪粪便、4%来自空气、2%来自河水、2%来自地下水、1%来自土壤。肠杆菌科(~56个ARGs)、莫拉菌科(~19个ARGs)、伯克氏菌科(~10个ARGs)、拟杆菌科(~6个ARGs)和链球菌科(~6个ARGs)是主要的ARGs宿主。它们主要存在于养殖村人肠道和养殖环境中,在非养殖村人肠道中极少检出。值得注意的是,肠杆菌科和莫拉菌科携带完全不同的ARGs种类。
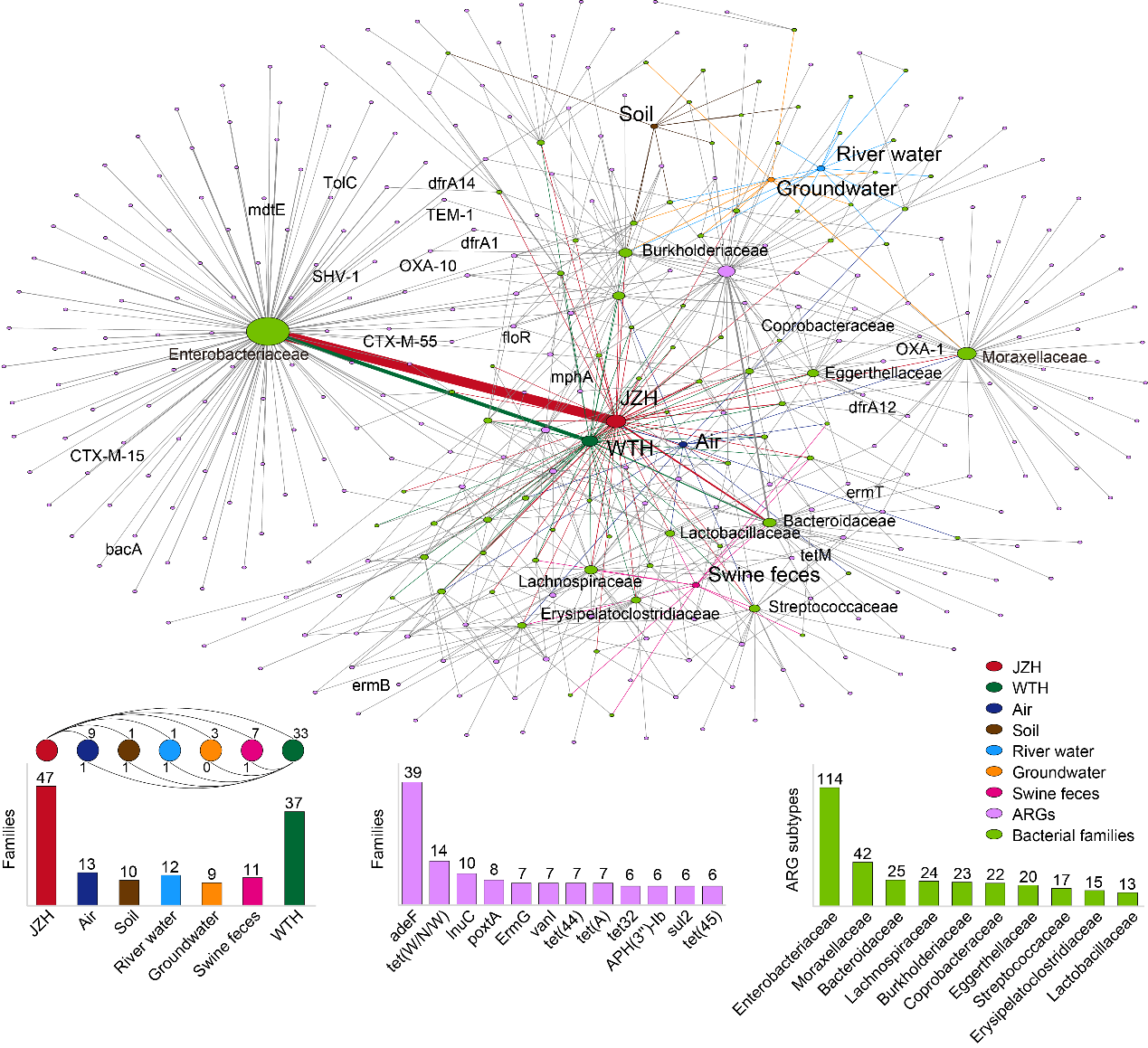
Fig. 5. Composition of MAGs-resolved ARGs in bacterial families. Names of the predominant families and “high-risk” ARGs are shown in the plot. Node size and edge thickness represent the degree and MAG number of each node, respectively. Values on bar plots are detected numbers of the families or ARGs in each group.
研究发现,大多数ARGs只存在于某个细菌科或属内,少数ARGs存在于不同的细菌门中。不论人肠道还是环境,同一个细菌属中的同种ARGs的序列更为保守,说明其具有相似的进化过程。
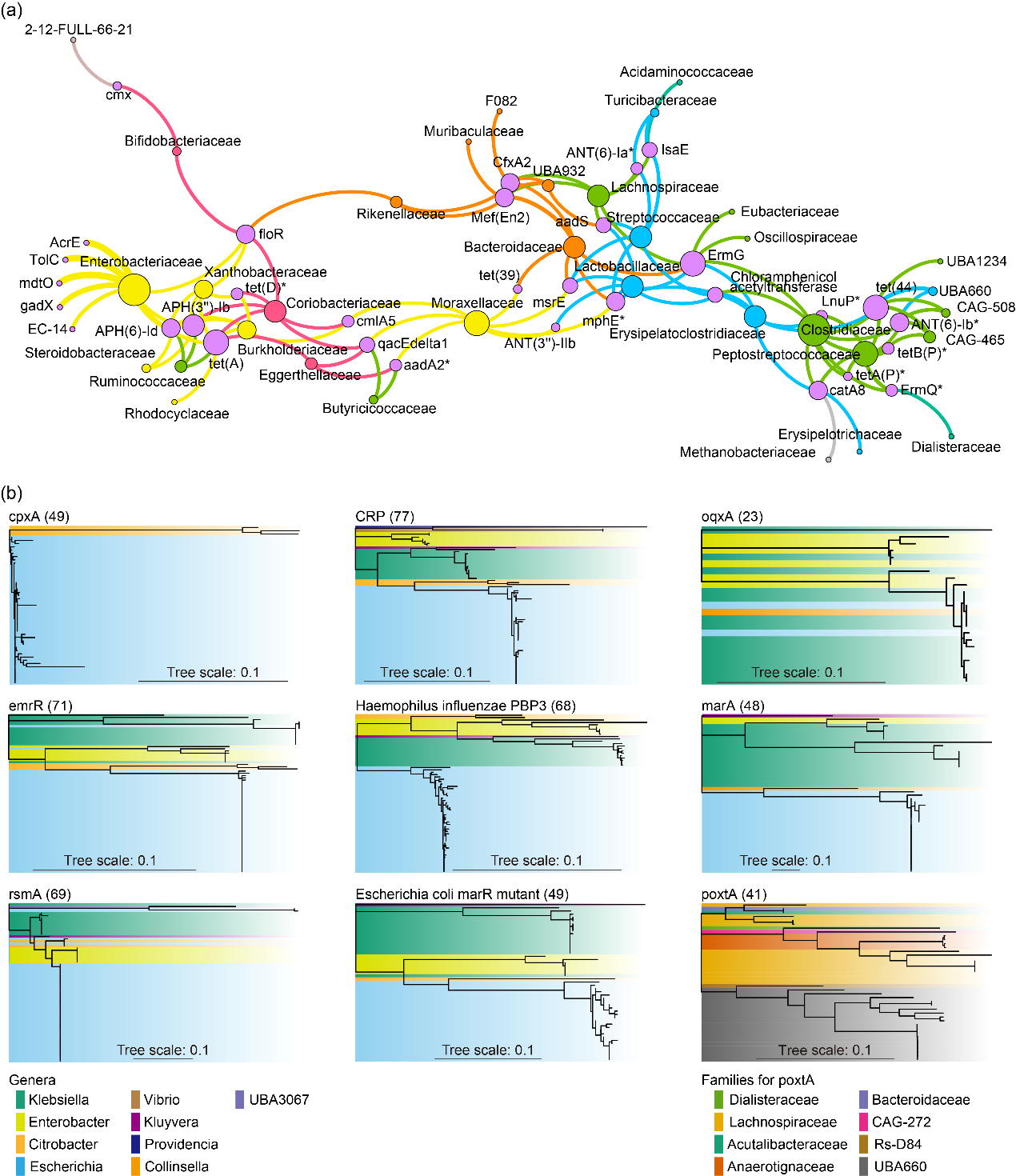
Fig. 6. Genomic ARGs’ homology in gut and environmental samples. (a) Bacterial hosts of highly conservative ARGs (the ARGs clustered into one group with 99% similarity and 95% coverage); (b) Phylogenetic trees of the moderately conservative ARGs (the ARGs clustered into multiple groups with 99% similarity and 95% coverage).
结论
本研究强调了养殖活动对当地居民肠道微生物群和抗性组的重要影响;肠道细菌耐药发展与养猪场环境暴露密切相关。与使用抗生素和改变生活环境相比,长期生活在这种环境可能会给肠道健康带来更多样和持久的改变,尽管其结果受到许多因素的影响,例如遗传,年龄、生理性别、个人卫生、饮食习惯、压力和职业。未来研究应力求阐明环境介质在ARGs和宿主细菌沿“动物-环境-人”传播的关键作用,并确定促进人肠道细菌耐药发展的主要因素。
致谢
本研究得到国家自然科学基金(NSFC 42030703、U1701242和42177226)、国家重点研发计划(2020YFC1806901)、中国博士后科学基金(2022M711215)以及广东省基础应用研究基金(2019A1515110131)的支持。同时感谢参与野外采样的团队成员和两个村委会的支持。
系列论文
Gao, FZ, He, LY, He, LX, et al. 2024. Swine farming shifted the gut antibiotic resistome of local people. J Hazard Mater, https://doi.org/10.1016/j.jhazmat.2023.133082.
Gao, FZ, He, LY, Chen, X, et al. 2023. Swine farm groundwater is a hidden hotspot for antibiotic-resistant pathogenic Acinetobacter. ISME Commun, https://doi.org/10.1038/s43705-023-00240-w.
Gao, FZ, He, LY, Bai H, et al. 2023. Airborne bacterial community and antibiotic resistome in the swine farming environment: Metagenomic insights into livestock relevance, pathogen hosts and public risks. Environ Int, https://doi.org/10.1016/j.envint.2023.107751.
Gao, FZ, He, LY, He, LX, et al. 2020. Untreated swine wastes changed antibiotic resistance and microbial community in the soils and impacted abundances of antibiotic resistance genes in the vegetables. Sci Total Environ, https://doi.org/10.1016/j.scitotenv.2020.140482.
Gao, FZ, Zou HY, Wu DL, et al. 2020. Swine farming elevated the proliferation of Acinetobacter with the prevalence of antibiotic resistance genes in the groundwater. Environ Int, https://doi.org/10.1016/j.envint.2020.105484.